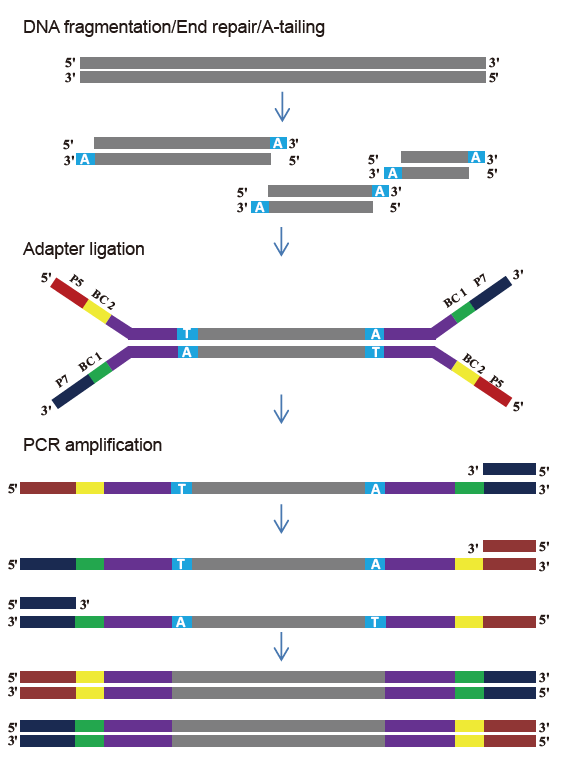
Комплект библиотеки TIANSeq DirectFast (иллюминация)
Функции
■ Хорошая однородность секвенирования: отсутствие систематической ошибки по основанию процесса фрагментации ДНК и процесса амплификации ПЦР.
■ Высокая эффективность преобразования библиотеки: создание высокоэффективной библиотеки может быть обеспечено для 1 нг образцов ДНК.
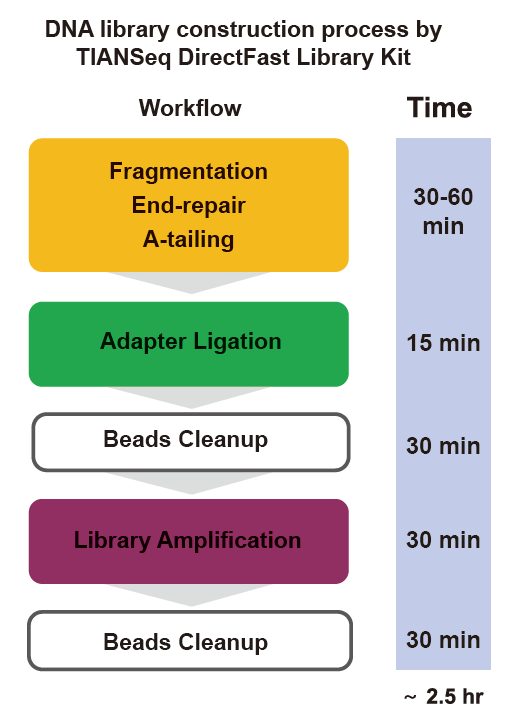
■ Быстрая работа: весь процесс создания библиотеки занимает всего 2,5 часа.
■ Рентабельность: не требуются специальные инструменты и оборудование。
Технические характеристики
Тип: Подготовка библиотеки ДНК для платформы высокопроизводительного секвенирования illumina
Образец: Геномная ДНК или большой фрагмент ДНК
Цель: Двухцепочечная ДНК
Начальный ввод образца: 1 нг - 1 мкг
Время операции: 2,5 часа
Последующие приложения: Секвенирование на платформе illumina
Все продукты могут быть настроены для ODM / OEM. Подробности см.пожалуйста, нажмите Индивидуальное обслуживание (ODM / OEM)
Гибкий ввод сэмплов и фрагментированный размер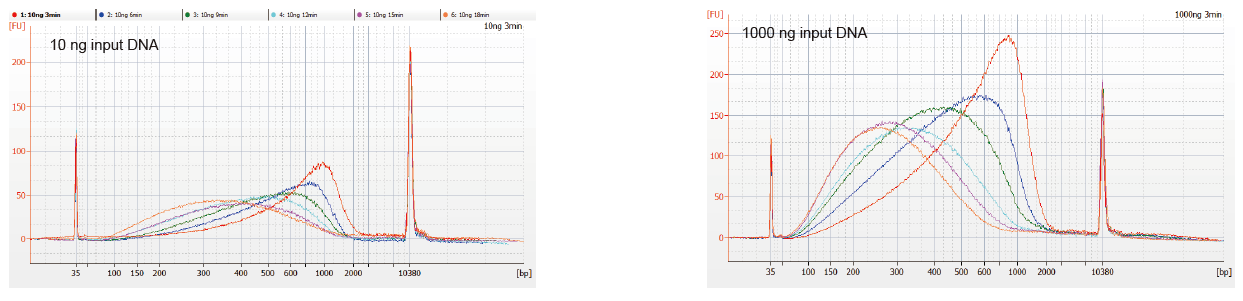 |
Рис. 1. Профили фрагментации ДНК при разном времени реакции. 10 нг и 1000 нг ДНК фрагментировали с использованием набора TIANSeq DirectFast DNA Library Kit. Продукты реакции, обработанные с разным временем реакции, очищали с помощью магнитных шариков 1,8 × Ampure XP и анализировали с помощью Angilent 2100. |
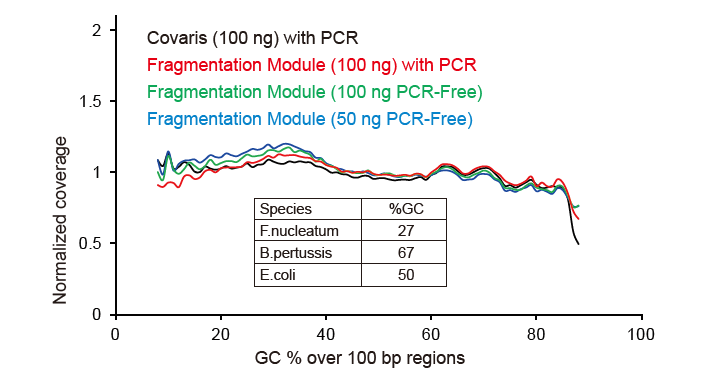
Покрытие Covaris-подобным секвенированием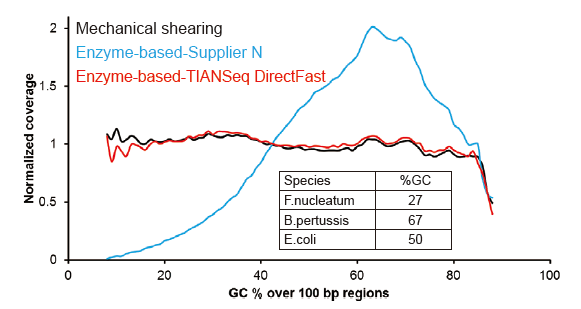 |
Рисунок 2. Сравнение покрытия генома различными методами подготовки библиотек. Три бактериальные геномные ДНК с различным содержанием GC смешаны эквимолярно, и результаты секвенирования охвата генома 100 нг смешанных библиотек ДНК с использованием этих методов были сравнены. Результаты показывают, что набор TIANSeq DirectFast Library Kit оказывает такое же влияние на фрагментацию ДНК, как и механический сдвиг, и отсутствует смещение по основанию для фрагментации. |
Отсутствие систематической погрешности при вводе всего 1 нг ДНК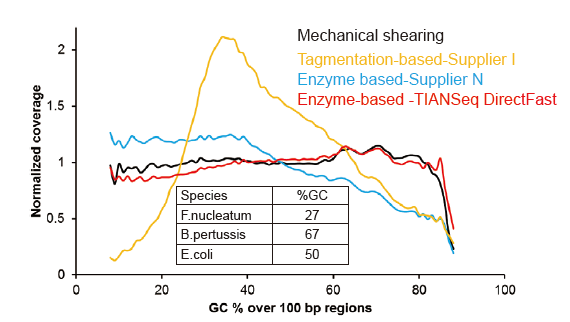 |
Рисунок 3. Сравнение покрытия генома различными методами подготовки библиотек. Три бактериальные геномные ДНК с различным содержанием GC смешаны эквимолярно, и результаты секвенирования покрытия генома 1 нг смешанных библиотек ДНК с использованием этих методов сравнивали. Результаты показывают, что набор TIANSeq DirectFast Library Kit обладает постоянным эффектом фрагментации с механическим сдвигом даже при вводе ДНК всего лишь 1 нг, и нет смещения основания. |
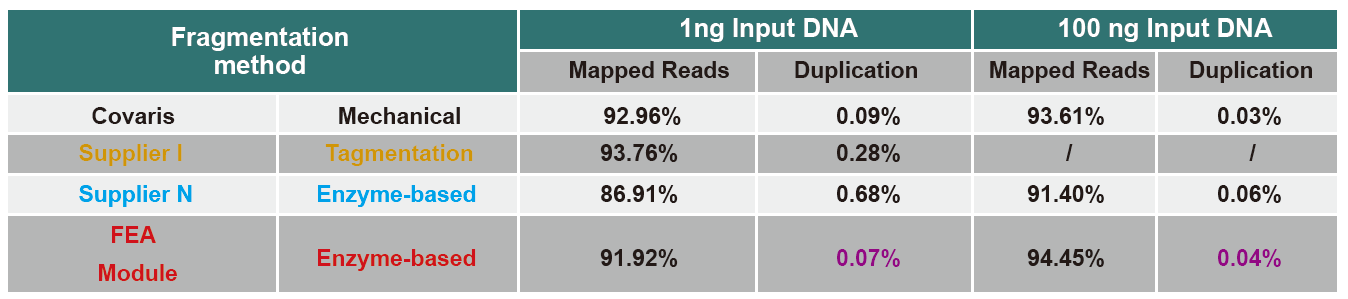
| Возможность работы без ПЦР
|
Рис. 4. Различный ввод геномной ДНК использовали для создания библиотеки с помощью ПЦР или конструирования библиотеки без ПЦР, и сравнивали результаты охвата генома. Результаты показывают, что при работе в одной пробирке и эффективных этапах построения библиотеки библиотека ДНК, созданная с помощью TIANSeq DirectFast Library Kit, сохраняет высокую согласованность с механическим сдвигом в распределении покрытия последовательности фрагментов для обоих рабочих процессов с обогащением ПЦР без ПЦР. |
Статистика эффективности и доходности строительства библиотеки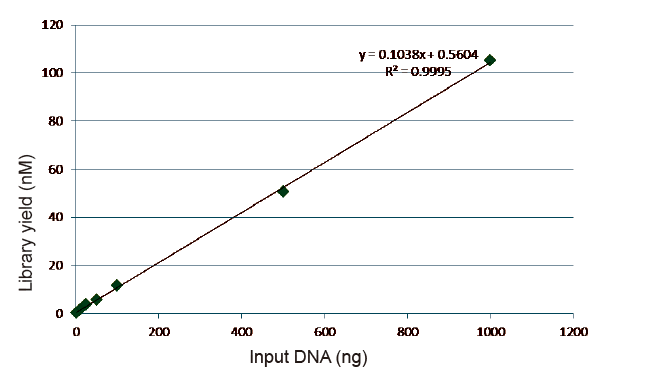 |
Рисунок 5. Результаты количественного анализа библиотеки ДНК, полученной с помощью кПЦР, после конструирования библиотеки методом без ПЦР для образцов с различными исходными количествами (1, 10, 25, 50, 100, 500, 1000 нг). Линейный регрессионный анализ показывает, что доходность библиотеки имеет хорошую линейную зависимость в широком диапазоне входных выборок. При вводе ДНК всего лишь 1 нг эффективность построения библиотеки не снижается. |
Сравнение данных секвенирования различных продуктов
В настоящее время технология высокопроизводительного секвенирования в основном основана на технологии секвенирования следующего поколения. Поскольку длина считывания технологии секвенирования следующего поколения ограничена, мы должны разбить последовательность полной длины на библиотеки небольших фрагментов для секвенирования. В соответствии с потребностями различных экспериментов по секвенированию мы обычно выбираем одностороннее или двустороннее секвенирование. В настоящее время фрагменты ДНК библиотеки секвенирования следующего поколения обычно распределяются в диапазоне 200-800 п.н.

а) ДНК плохого качества и содержит ингибиторы. Используйте высококачественные образцы ДНК, чтобы избежать подавления активности ферментов.
б) Количество образца ДНК недостаточное при использовании метода без ПЦР для создания библиотеки ДНК. Когда ввод фрагментированной ДНК превышает 50 нг, рабочий процесс без ПЦР может выборочно выполняться в процессе создания библиотеки. Если число копий библиотеки слишком мало для прямого секвенирования, библиотеку ДНК можно амплифицировать с помощью ПЦР после лигирования адаптера.
c) Загрязнение РНК приводит к неточному первоначальному количественному определению ДНК. Загрязнение РНК может присутствовать в процессе очистки геномной ДНК, что может привести к неточному количественному определению ДНК и недостаточной загрузке ДНК во время создания библиотеки. РНК можно удалить обработкой РНКазой.
А-1
а) Появляются маленькие фрагменты (60-120 пар оснований). Маленькие фрагменты обычно представляют собой адаптерные фрагменты или димеры, образованные адаптерами. Очистка с помощью магнитных шариков Agencourt AMPure XP может эффективно удалить эти фрагменты адаптера и обеспечить качество секвенирования.
б) Большие фрагменты появляются в библиотеке после ПЦР-амплификации. Размер ДНК-фрагмента библиотеки увеличится на 120 п.н. после лигирования адаптера. Если после лигирования адаптера фрагмент ДНК увеличивается более чем на 120 п.н., это может быть вызвано аномальной амплификацией фрагмента или чрезмерной амплификацией ПЦР. Избежать ситуации может уменьшение количества циклов ПЦР.
в) Аномальный размер фрагментов ДНК библиотеки после лигирования адаптера. Длина адаптера в этом наборе составляет 60 п.н. Когда два конца фрагмента лигируются с адаптерами, длина увеличивается только на 120 п.н. При использовании адаптера, отличного от того, который входит в этот комплект, свяжитесь с поставщиком, чтобы предоставить соответствующую информацию, например длину адаптера. Убедитесь, что рабочий процесс и работа эксперимента соответствуют шагам, описанным в руководстве.
г) Аномальный размер фрагмента ДНК до лигирования адаптера. Причина этой проблемы может быть вызвана неправильными условиями реакции во время фрагментации ДНК. Для разных вводов ДНК следует использовать разное время реакции. Если ввод ДНК превышает 10 нг, мы рекомендуем выбрать время реакции 12 мин в качестве начального времени для оптимизации, а размер фрагмента, произведенного в это время, находится в основном в диапазоне 300-500 п.н. Пользователи могут увеличивать или уменьшать длину фрагментов ДНК на 2-4 мин в соответствии со своими требованиями, чтобы оптимизировать фрагменты ДНК до необходимого размера.
А-2
a) Время фрагментации не оптимизировано. Если фрагментированная ДНК слишком мала или слишком велика, обратитесь к Руководству по выбору времени фрагментации, приведенному в инструкции, чтобы определить время реакции, и используйте эту временную точку в качестве контроля, дополнительно установите система реакции для продления или сокращения 3 мин для более точной настройки времени фрагментации.
А-3
Аномальное распределение ДНК по размеру после обработки фрагментацией
a) Неправильный метод оттаивания реагента для фрагментации или реагент не полностью перемешан после оттаивания. Разморозьте реагент 5-кратной смеси ферментов для фрагментации на льду. После размораживания равномерно перемешайте реагент, осторожно постучав по дну пробирки. Не перемешивайте реагент на вортексе!
б) Входной образец ДНК содержит ЭДТА или другие загрязнители. Истощение солевых ионов и хелатирующих агентов на этапе очистки ДНК особенно важно для успеха эксперимента. Если ДНК растворяется в 1 × TE, используйте метод, указанный в инструкции, для выполнения фрагментации. Если концентрация ЭДТА в растворе не определена, рекомендуется очистить ДНК и растворить ее в деионизированной воде для последующей реакции.
c) Неточная первоначальная количественная оценка ДНК. Размер фрагментированной ДНК тесно связан с количеством введенной ДНК. Перед обработкой фрагментацией необходимо точное количественное определение ДНК с использованием Qubit, Picogreen и других методов для определения точного количества ДНК в реакционной системе.
г) Подготовка реакционной системы не соответствует инструкциям. Подготовка фрагментированной реакционной системы должна проводиться на льду в строгом соответствии с инструкциями. Для обеспечения наилучшего эффекта все реакционные компоненты должны быть помещены на лед, а подготовка реакционной системы должна выполняться после полного охлаждения. После того, как приготовление будет завершено, нажмите или пипеткой, чтобы тщательно перемешать. Не перемешивайте!
1. Неправильный метод смешивания (вихрь, сильные колебания и т. Д.) Вызовет ненормальное распределение фрагментов библиотеки (как показано на следующем рисунке), что повлияет на качество библиотеки. Поэтому при приготовлении реакционного раствора смеси для фрагментации осторожно пипеткой поднимайте и опускайте, чтобы перемешать, или кончиком пальца встряхните и равномерно перемешайте. Будьте осторожны, не смешайте с вихрем.

2. Для создания библиотеки необходимо использовать ДНК высокой чистоты.
■ Хорошая целостность ДНК: полоса электрофореза составляет более 30 т.п.н., без хвостов.
■ OD260 / 230:> 1,5
■ OD260 / 280: 1,7–1,9
3. Количество вводимой ДНК должно быть точным. Для количественной оценки ДНК рекомендуется использовать методы Qubit и PicoGreen, а не Nanodrop.
4. Необходимо определить содержание ЭДТА в растворе ДНК. ЭДТА имеет большое влияние на реакцию фрагментации. Если содержание ЭДТА высокое, перед последующим тестом необходимо провести очистку ДНК.
5. Раствор для реакции фрагментации должен быть приготовлен на льду. Процесс фрагментации чувствителен к температуре и времени реакции (особенно после добавления усилителя). Чтобы обеспечить точность времени реакции, приготовьте реакционную систему на льду.
6. Время реакции фрагментации должно быть точным. Время реакции этапа фрагментации будет напрямую влиять на размер продуктов фрагмента, таким образом влияя на распределение по размеру фрагментов ДНК в библиотеке.
1. Какой тип образца применим к этому набору?
Применимый тип образца этого набора может быть тотальной РНК или очищенной мРНК с хорошей целостностью РНК. Если для создания библиотеки используется полная РНК, рекомендуется сначала использовать набор для истощения рРНК (каталожный номер 4992363/4992364/4992391) для удаления рРНК.
2. Можно ли использовать образцы FFPE для создания библиотеки с помощью этого набора?
МРНК в образцах FFPE будет деградировать до определенной степени с относительно плохой целостностью. При использовании этого набора для построения библиотеки рекомендуется оптимизировать время фрагментации (сократить время фрагментации или не выполнять фрагментацию).
3. При использовании шага выбора размера, приведенного в руководстве по продукту, что может вызвать небольшое отклонение вставленного сегмента?
Выбор размера должен осуществляться в строгом соответствии с этапом выбора размера, описанным в данном руководстве по продукту. Если есть отклонение, причина может заключаться в том, что магнитные шарики не сбалансированы до комнатной температуры или не полностью перемешаны, пипетка неточная или жидкость осталась в наконечнике. Для эксперимента рекомендуется использовать насадки с низкой адсорбцией.
4. Подбор переходников в библиотечном строительстве.
Конструктор библиотеки не содержит реагента-адаптера, и рекомендуется использовать этот набор вместе с адаптером TIANSeq Single-Index (Illumina) (4992641/4992642/4992378).
5. Контроль качества библиотеки
Количественное определение библиотеки: Qubit и qPCR используются для определения массовой и молярной концентрации библиотеки соответственно. Операция выполняется строго в соответствии с руководством по продукту. Концентрация библиотеки обычно соответствует требованиям секвенирования NGS. Определение диапазона распределения библиотеки: использование Agilent 2100 Bioanalyzer для определения диапазона распределения библиотеки.
6. Выбор номера цикла амплификации.
Согласно инструкциям, количество циклов ПЦР составляет 6-12, и количество необходимых циклов ПЦР следует выбирать в соответствии с вводом образца. В библиотеках с высоким выходом избыточная амплификация обычно происходит в той или иной степени, что проявляется немного большим пиком после пика целевого диапазона при обнаружении Agilent 2100 Bioanalyzer, или обнаруженная концентрация Qubit ниже, чем у qPCR. Небольшая избыточная амплификация - нормальное явление, которое не влияет на секвенирование библиотеки и последующий анализ данных.
7. В профиле обнаружения биоанализатора Agilent 2100 появляются всплески.
Появление всплесков при обнаружении Agilent 2100 Bioanalyzer связано с неравномерной фрагментацией образцов, где будет больше фрагментов определенного размера, и это станет более очевидным после обогащения ПЦР. В этом случае рекомендуется не выполнять выбор размера, то есть установить условие фрагментации на 94 ° C в течение 15 мин инкубирования, когда распределение фрагментов небольшое и концентрированное, а гомогенность может быть улучшена.
Категории товаров
ПОЧЕМУ ВЫБРАЛИ НАС
С момента своего основания наша фабрика разрабатывает первоклассные продукты с соблюдением принципа
качества в первую очередь. Наша продукция завоевала отличную репутацию в отрасли и пользуется доверием среди новых и старых клиентов.